MODAPLEX Platform
Simplify Molecular Precision Testing
MODAPLEX Solution
Leverage MODAPLEX to Improve Your Multi-Gene Testing Workflow

In an era where healthcare is advancing at an unprecedented pace, there is a growing need for sophisticated technologies to establish multi-gene testing as the new standard of care. This approach not only maximizes the utilization of limited tissue samples but also significantly reduces critical turnaround times. The MODAPLEX platform’s 4-hour automated workflow enables hospitals and pathology facilities to adopt this valuable technology in-house, promising faster results and improved care coordination.
MODAPLEX Platform
MORE IN ONE
MODAPLEX – MORE in ONE
More actionable insights for improved decision-making.


MODAPLEX – MORE in ONE
More actionable insights for improved decision-making.

MORE MODALITIES
Perform multiparametric analysis through parallel detection of mutations, copy number variations, gene expressions, gene fusions, and more.
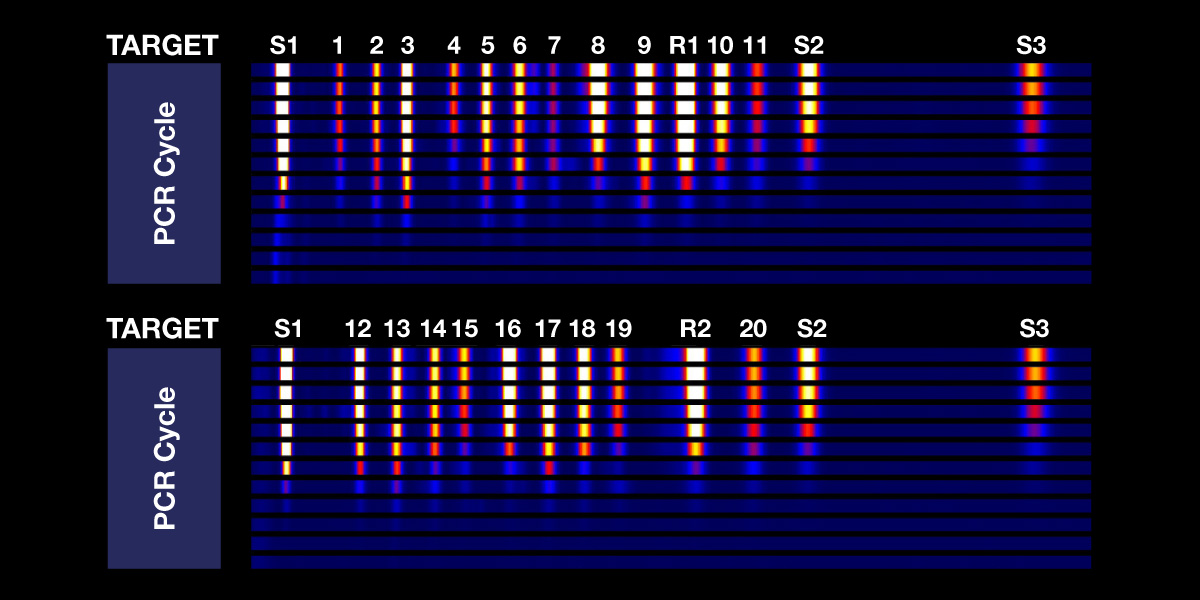
MORE MULTIPLEXING
Harness the power of multiplexing to efficiently detect multiple targets in a single well and benefit from assays designed to uncover all the key genetic alterations in a single workflow.

MORE MULTIPLEXING
Harness the power of multiplexing to efficiently detect multiple targets in a single well and benefit from assays designed to uncover all the key genetic alterations in a single workflow.

MORE FLEXIBILITY
Simultaneously analyze up to 48 wells with any sample and marker combination using the universal PCR program uniquely tailored to all MODAPLEX assays.
MORE SPEED
Setting up a MODAPLEX analysis is as straightforward as setting up a PCR. The MODAPLEX load- and walk-away workflow is identical for all assays and provides results within 4 hours.


MORE SPEED
Setting up a MODAPLEX analysis is as straightforward as setting up a PCR. The MODAPLEX load- and walk-away workflow is identical for all assays and provides results within 4 hours.

MORE SIMPLICITY
Analyzing your results only takes minutes with the intuitive MODAPLEX Reporter software which generates clear reports for each MODAPLEX assay.
MODAPLEX Enabling Platform
A Comprehensive Molecular Profiling Solution for Actionable Biomarker Panels
MODAPLEX for Molecular Profiling
Assays Provided by BIOTYPE
Use the MODAPLEX instrument with ready-to-use molecular test solutions that overcome current multiplexing constraints.

Upgrade your assay portfolio
BIOTYPE offers you the possibility to develop, transfer, and commercialize your multi-gene & multi-marker assays on the MODAPLEX.
MODAPLEX for Assay Development & Licensing
MODAPLEX for Assay Development & Licensing
Upgrade your assay portfolio
BIOTYPE offers you the possibility to develop, transfer, and commercialize your multi-gene & multi-marker assays on the MODAPLEX.
Contact
BIOTYPE GmbH
Moritzburger Weg 67
01109 Dresden
Germany
Mail: modaplex@biotype.de
Tel: +49 351 8838 400
